行业新闻
Python的遗传算法GA优化深度置信网络DBN超参数回归预测
DBN超参数众多,包括隐含层层数、各层节点数、无监督预训练阶段的训练次数及其学习率、微调阶段的训练次数及其学习率、与Batchsize,如果采用SGD相关优化器,还有动量项这个超参数。总之就是特别多,手动选择的话很难选到最佳超参数组合,为此采用遗传算法对上述超参数进行优化。
之前写过MATLAB版本的DBN超参数优化,今天这个是python/torch版本的DBN超参数优化。具体运行环境python36、torch1.2。
? ? ? ? 话不多说,直接上结果。数据结构是多输入单输出,按照7:3随机划分训练集与测试集。
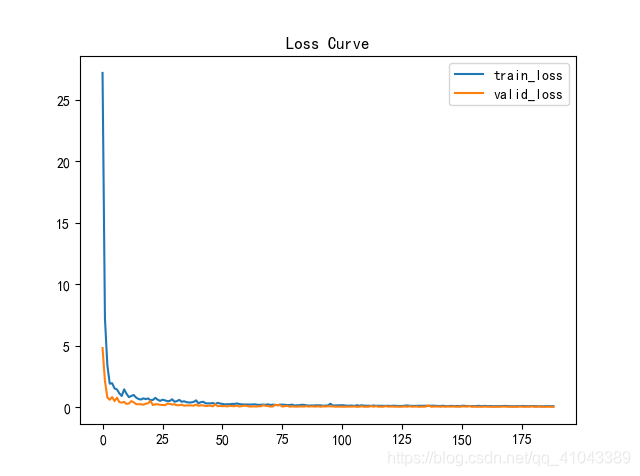
1、DBN预测,代码如下
结果如下:
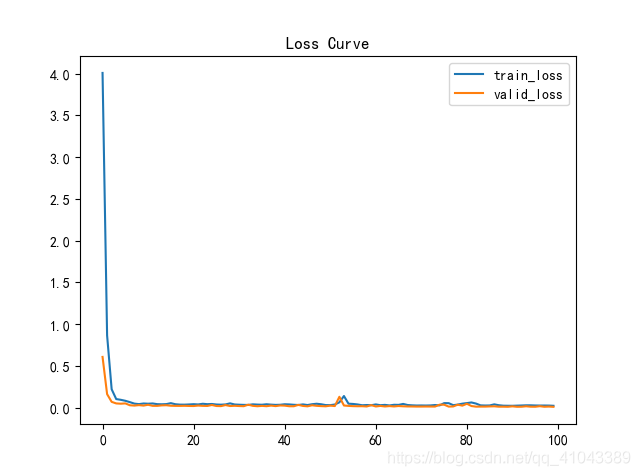
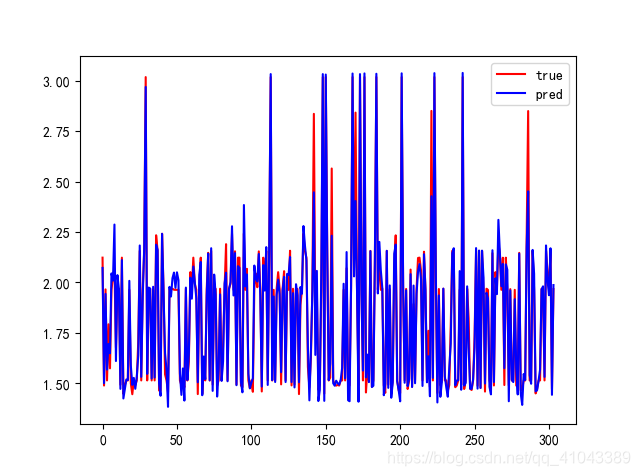
指标如下:
mape: 0.02131540499483512 ?
rmse: 0.06900334093296646 ?
mae: 0.04012139316438816 ?
R2: 0.9663487401772392
2、遗传优化DBN超参数
本文利用GA对DBN的超参数进行寻优,以最小化DBN预测值与实际值的MSE为适应度函数,目的就是通过GA找到一组超参数,用这组超参数训练的DBN,具有最小的网络误差。寻优参数及其范围设置如下:
操作要点:
1)前3个超参数是浮点型、后面的都是整型
2)遗传算法每条DNA的长度是10,就是前7个参数是两个学习率、动量项、两个训练迭代次数、batchsize与层数,因为我们程序设置的最大隐含层是3(ub中是4,是因为random.int这个函数是左闭右开),所以一共10维,即使某DNA中层数是2,也是10维,只不过最后1层为0,例如:
[0.1,0.5,0.9,10,25,64,1,10,0,0],则代表两个学习率分别为0.1 0.5;动量为0.9; 两个迭代次数分别是10,25,batchsize是64,隐含层层数是1,神经元为[10]
[0.1,0.5,0.9,10,25,64,2,10,15,0],则代表两个学习率分别为0.1 0.5;动量为0.9; 两个迭代次数分别是10,25,batchsize是64,隐含层层数是2,各层神经元分别为[10,15]
[0.1,0.5,0.9,10,25,64,5,10,15,20],则代表两个学习率分别为0.1 0.5;动量为0.9; 两个迭代次数分别是10,25,batchsize是64,隐含层层数是3,各层神经元分别为[10,15,30]
当然哈,ub中这个参数是可调的,随便设都行,只要大于1。
3)交叉操作的时候,隐含层层数这个参数不交叉,因为两个DNA如果层数不一致,交叉了的话,后面的节点数就不对了。如果要交叉这个参数的话,就把后面各层的节点数一起进行交叉。
4)变异的时候,隐含层层数发生改变之后,先把该条DNA的各层节点数全部置0 ,然后重新根据层数进行赋值。
寻优结果适应度曲线如图所示。
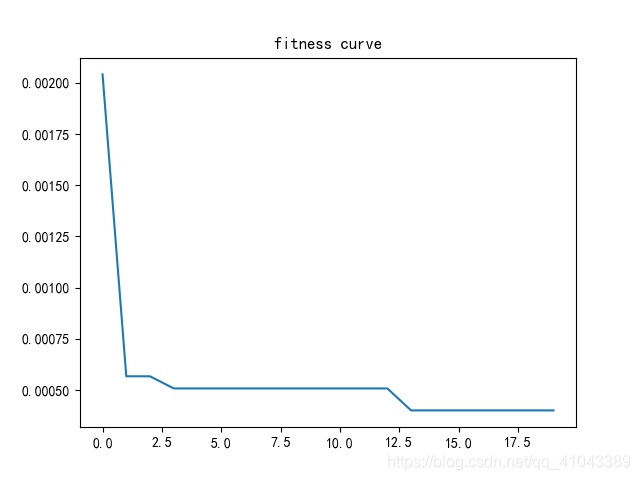
?对应GA每代优化后的最优超参数组合如下表所示。可见最优DBN的隐含层层数为3层,各层的节点数是92、35、54。
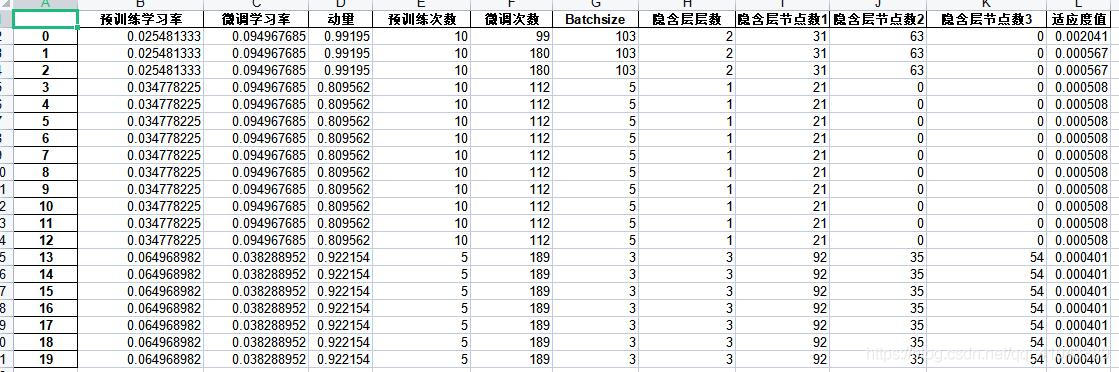
?取最后一代优化得到的最优超参数组合(就是第9行那个)用来训练DBN,得到的结果如下:
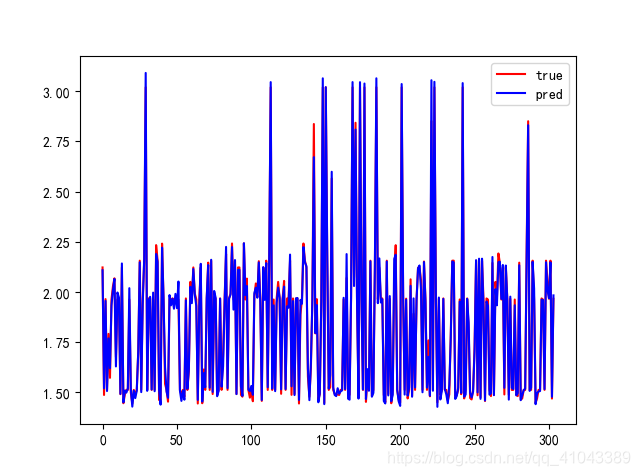
3、结果对比
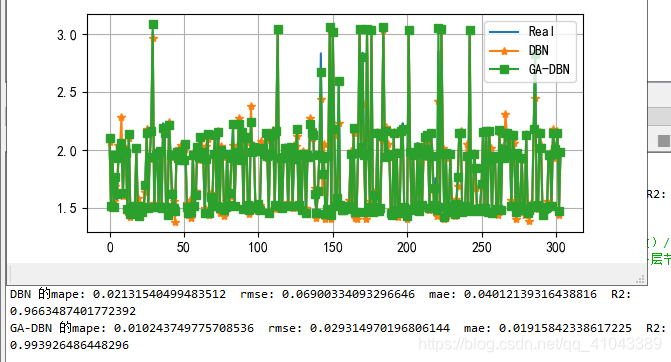
开丰新闻
联系我们
公司名称: 开丰娱乐-开丰五金配件机电公司
手 机: 13800000000
电 话: 400-123-4567
邮 箱: admin@youweb.com
地 址: 广东省广州市天河区88号

